植物代谢组学作为一项新兴的组学技术,在代谢途径解析、基因功能解析以及作物品种改良等方面得到了广泛应用,展现出了极大的潜力。伴随质谱技术的进步以及检测成本的下降,对于群体规模的植物代谢组学检测需求与日俱增。但是,针对大样本量的代谢组学数据处理以及数据挖掘的流程开发却相对迟缓,现有的流程多数由各个实验室自主搭建,并且要求使用者具备一定的编程经验。所以,开发出一款对用户友好的大规模植物代谢组学数据分析流程有着重要意义。
近日张学斌教授团队在Journal of Integrative Plant Biology期刊上发表了以“MetMiner: A user-friendly pipeline for large-scale plant metabolomics data analysis”为题的论文 (https://doi.org/10.1111/jipb.13774),介绍了一款用户友好的,专为大规模植物代谢组学数据处理及数据挖掘打造的分析流程MetMiner。

MetMiner 分析流程由三个部分组成:其一为上游数据处理,其二为下游数据分析,其三为进阶数据挖掘。整体是基于 R-shiny 进行开发的,拥有用户友好且交互性强的图形界面,只需通过数据上传、参数选择等简便步骤,就能完成完整的植物代谢组学数据分析与数据挖掘工作。
上游数据处理的目的是把 LC-MS 原始数据经由数据清洗、标准化等流程转变为能够用于下游统计分析的干净数据。这一部分调用了 tidyMass framwork(Shen et al. 2022),先进的质谱数据存储和管理方式不但确保了 MetMiner能够从不同种类的质谱数据展开分析,还保障了数据分析的透明性、可追溯性以及可重复性。
下游数据分析涵盖了代谢物的注释、分类、基础统计分析以及富集分析,此研究有针对性地开发了代谢组下游分析工具包(MDAtoolkits)。MDAtoolkits 内部设置了六个针对植物的特异MS2代谢物数据库以及三个MS1代谢物数据库,保证了植物代谢组学中代谢物注释的准确性。另外,MDAtoolkits整合了多元统计分析和单元统计分析,能够迅速鉴定差异代谢物,并且基于 ClassyFire数据库和KEGG数据库开发了代谢物分类富集分析和通路富集分析,以辅助用户解析差异代谢物潜在的生物学功能。
在复杂的代谢组学实验设计中,标志代谢物的挖掘工作相对较为困难,本研究提出了迭代WGCNA的策略,通过多轮WGCNA把无法聚类的代谢物逐步排除掉,最终将具有相同积累规律的代谢物归到不同的模块,构建代谢物共积累网络。通过对模块进行通路富集分析、分类富集分析以及提取枢纽代谢物(hub metabolite),从而快速锁定标志代谢物。
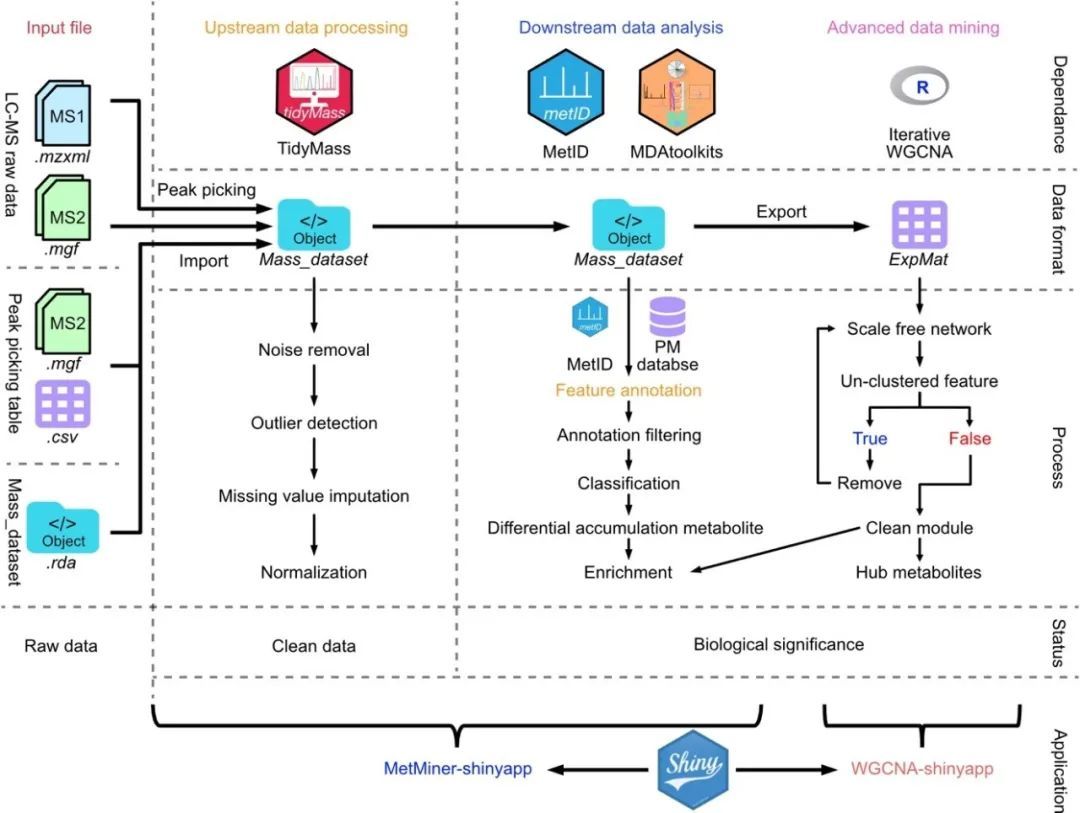
图1. MetMiner流程结构
MetMiner能够通过Shiny Server部署在高性能服务器或者计算集群上,调用更多的计算资源来达成对大样本量的代谢组学数据分析。此外,MetMiner的设计理念充分考量了流程的“易用性”和“交互性”。“Resuming analysis from the unfinished steps”这一功能使用户能够随时继续未完成的分析,或者有选择地针对特定步骤重新进行分析。“Interactive plot”提供了强大的图表数据交互能力,让用户能直接从图片中获取相应信息。例如在图2中,通过点击差异分析火山图中代表差异代谢物的点,该代谢物的统计分析结果、代谢物注释、MS2 spactra以及化学结构图就会相应地显示出来。这简化了用户对数据的提取操作,提高了数据分析及信息获取的效率。
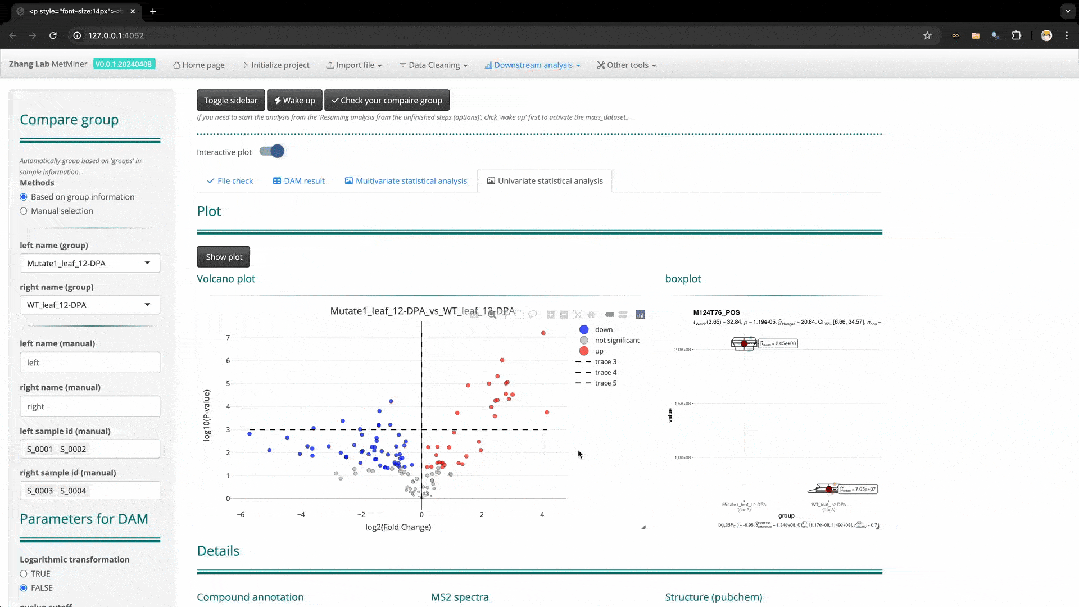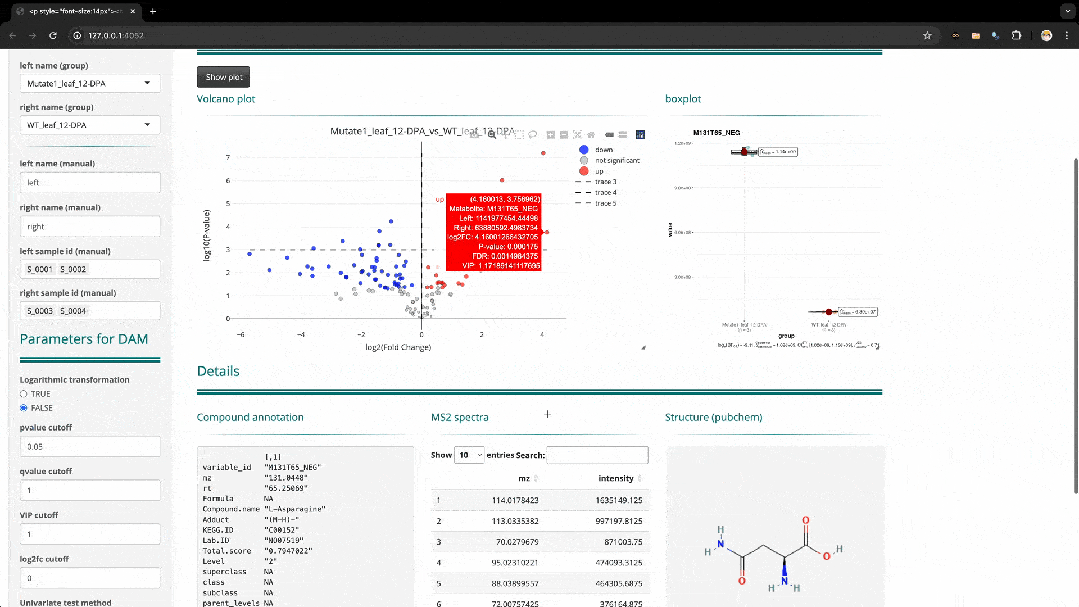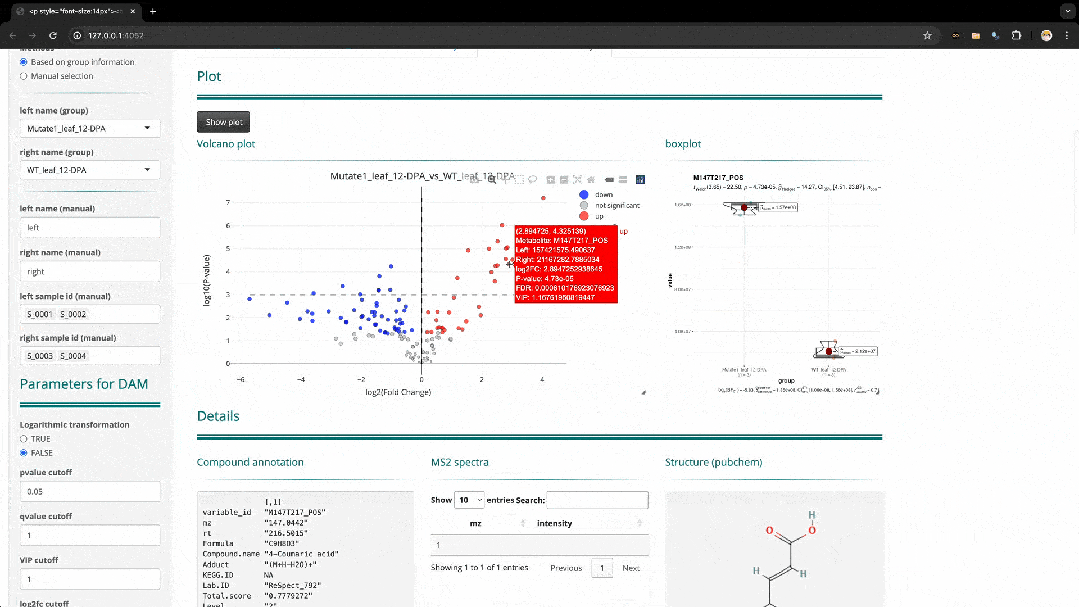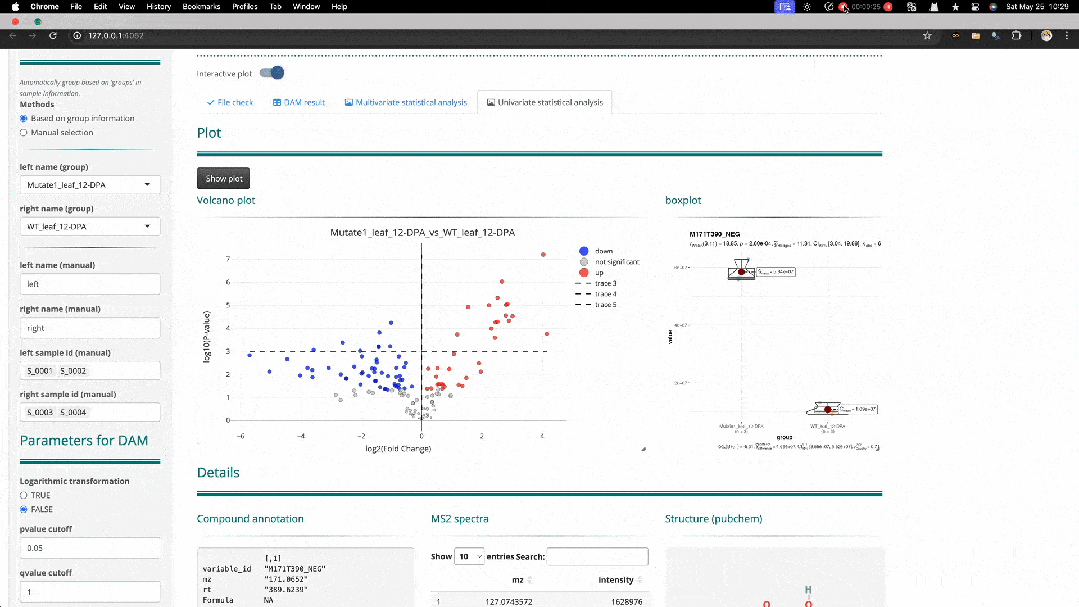
图2. 图形交互展示
最后,该研究运用MetMiner流程对206份拟南芥 F-box 突变体群体拟靶代谢组学数据展开了深入剖析,迅速锁定了标志代谢物硫代葡萄糖苷(glucoraphanin)和芥子酰苹果酸(sinapoyl malate),并且通过靶向检测证实了 MAX2突变致使硫代葡萄糖苷增多,KFB20突变致使芥子酰苹果酸增多,这一结果表明了MetMiner在群体规模代谢组学数据挖掘中的准确性与高效性。另外,由MetMiner处理的不同质谱平台生成的非靶代谢组学数据,在代谢物定性及定量方面呈现出极高的一致性,这意味着MetMiner在代谢物注释方面具备较强的稳健性。
河南大学省部共建作物逆境适应与改良国家重点实验室博士后王骁、已毕业硕士生梁爽、青年教师杨文奇以及青年教师禹坷为该论文的共同第一作者,张学斌教授、青年教师张君丽为该论文的共同通讯作者。本研究得到国家重点研发计划、河南省高等学校重点科研项目、国家自然科学基金、河南省博士后科研资助项目以及Biotechnology and Biological Sciences Research Council (BBSRC, UK) “A China-UK joint phenomics consortium to dissect the basis of crop stress resistance in the face of climate change”的资助。